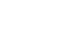水稻,作為是世界上重要的糧食作物之一,為全世界超過35億人的主食,維系超過10億人的生計。因此,稻米對全球糧食安全至關重要,提升稻米生產系統的效率是應對糧食安全挑戰的關鍵一環。野生近緣種為水稻提供了寶貴的遺傳資源。生長于西非熱帶地區的長雄野生稻(又稱非洲野生稻,Oryza longistaminata)表現出多年生生長和極高的生物量生產能力,相關有益等位基因已經被轉移到商業水稻品種中。除了對育種的貢獻外,長雄野生稻也是研究根莖遺傳基礎和發育方面的重要研究對象。
長雄野生稻 (2x=2n=12) 主要生長在西非的熱帶地區,是一種AA基因組型,主要生長在靠近淡水資源和沼澤的地區。雖然很少用于人類食用,但該物種具有抵抗力高、根莖無性繁殖和生物脅迫等多種有益的特性。由于測序技術限制和基因組復雜的組織結構,此前的非洲野生稻的參考基因組中仍然存在未被充分展現的復雜區域,這限制了對其開展深入且詳盡的研究工作。
為解決這一問題,近日,華大研究院聯合云南大學,依托華大序風的CycloneSEQ納米孔測序平臺,完成長雄野生稻343 Mb的端粒到端粒(T2T)的基因組組裝,涵蓋了12條染色體上的所有端粒和著絲粒,新組裝的基因組比以前版本有了明顯的改進,為栽培稻野生近緣種中有益等位基因的探索和開發提供了寶貴的資源。
基因組組裝
對培育的長雄野生稻嫩葉提取DNA后進行測序,獲得25.6 Gb CycloneSEQ超長序列數據,21 Gb MGI-Seq雙端測序數據,27.3 Gb PacBio HiFi數據,32 Gb Hi-C數據。使用K-mer評估基因組大小為357 Mb,雜合度為1.27%。混合組裝首先獲得了一個343 Mb基因組,contig N50為26.02Mb,隨后使用Hi-C數據將組裝序列合并為12個假染色體,使用TGS-gapcloser填補剩余空白。利用端粒重復序列鑒定到基因組全部的24個端粒,鑒定到染色體的著絲粒區域長度在0.3 Mb至1.8 Mb之間。在組裝基因組準確性和完整度評估方面,雙端測序數據比對率達97.27%,BUSCO分析完整度達到98.6%,LTR組裝指數(LAI)為20.71(符合參考基因組金標準),Merqury組裝質量QV值達到52.08(即堿基準確率高于99.999%)。與之前已發表的長雄野生稻基因組(Reuscher et al., 2018)進行編碼基因共線性比較分析,在基因組范圍鑒定出28,627個共線性編碼基因,與預期一致,表明組裝的T2T基因組與已發表的基因組具有高度一致性。

基因組注釋
使用從頭分析和同源比較分析,在基因組中鑒定出134 Mb重復序列,大約占全基因組的40.73%。重復序列在12個染色體和全基因組水平上高度一致。LTR和DNA轉座子為主要的重復單元,分別占據大約20.9%和18.5%。重復水平達到中度,與其他稻屬的其他基因組相近。基因組的著絲粒由于其高度重復,組裝難度較高。本次組裝的T2T基因組發現著絲粒區域飽含轉座元件并只含有少量基因。著絲粒區域中, LTR中大部分為Gypsy元件。
基因組中預測出有33,177個編碼基因,平均長度2,439 bp,平均編碼序列長度達到1,138 bp。功能分析顯示95.74%的編碼基因可在蛋白公共數據庫被注釋,展示了基因預測的準確性高。
長雄野生稻的T2T基因組組裝
(從外到內:GC含量、蛋白編碼基因、重復序列、LTR-Gypsy、HTR-Copia、共線性區塊)
基因組結構變異
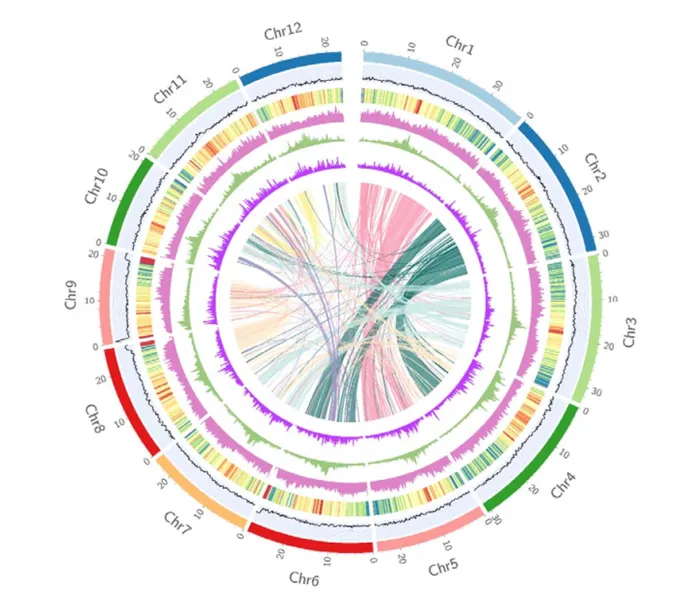
栽培稻與長雄野生稻的全基因組結構變異推測發現,兩個基因組間包含3,738,150個SNP位點,204個倒置區塊,11,706個重復區域,11,175個倒置重復,3,077個移位和3,015個倒置移位。超105 Mb的結構變異顯示出兩個物種間的巨大差異。GO分析結構變異相關基因展示出與催化活性、嘌呤核糖核苷酸結合、腺苷核糖核苷酸結合和端粒維持的相關性。
栽培稻與長雄野生稻T2T基因組的共線性分析和變異分析
(Reference基因組為 O. sativia,Query基因組為 O. longistaminata)
基因組片段重復分析
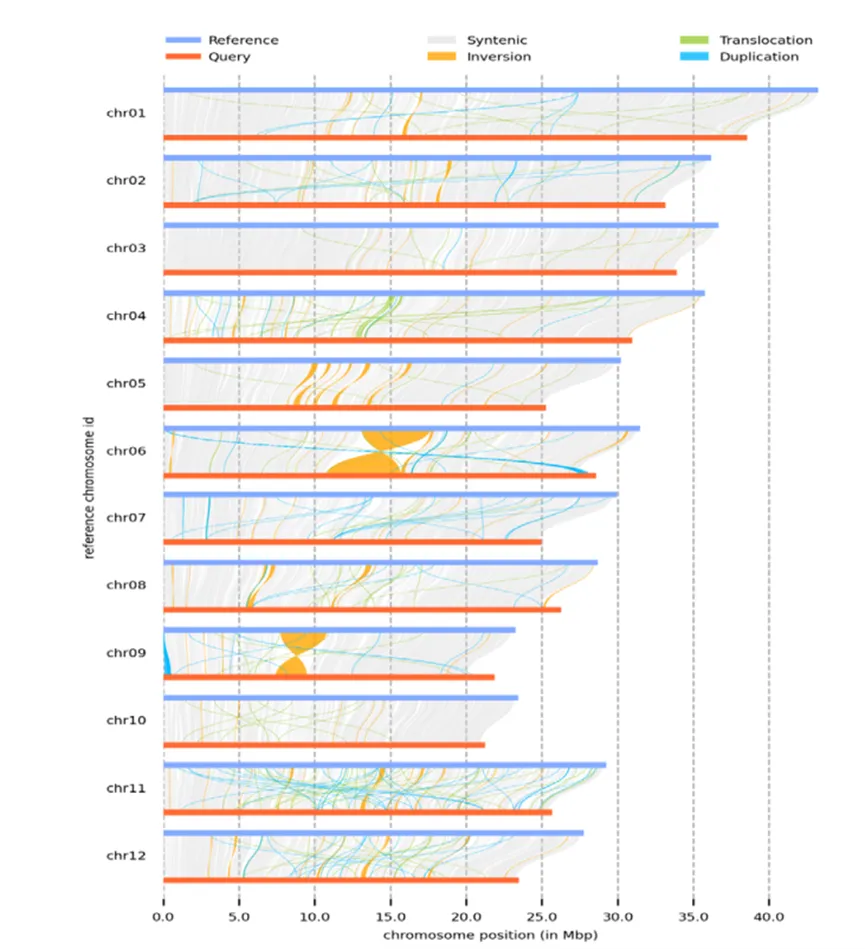
片段重復(Segmental Duplications, SDs)是指基因組中大于1 Kb的至少有90%序列一致性的重復片段。SD中常含大量重復基因,在基因創新中有重要作用。先前基因組中不準確的SD注釋限制了基因組結構和進化的理解。長雄野生稻的T2T基因組提供了SD研究更準確的參考信息。使用BISER工具一共鑒定出30.2 Mb的SD,并發現在基因組水平并非均勻分布。SD在1/4/3/2號染色體中含量更高,在9/10/5號染色體中更少。該不均勻分布提示1/4/3/2號染色體可能對水稻進化中的作用方式為先前未知的。
使用BLASTP在SD區域鑒定重復基因,一共發現4,179對同源基因,1,233對為高度匹配,并發現大部分SD是近期發生(Ks=0.3)。GO分析顯示這些等位基因與細胞氨基酸代謝、羧酸代謝和輔因子結合相關。
長雄野生稻基因組的片段復制分析
NBS基因家族和轉錄因子
核苷酸結合位點-亮氨酸富集重復單元(NBS-LRR)蛋白為植物對抗病原體的抗性蛋白的最大的家族。11種水稻的NBS-LRR分析發現,長雄野生稻有654 NBS-LRR基因,比其他物種基因更少,即抗性基因更少。說明長雄野生稻對病原體的識別和免疫識別的能力進化。
稻屬內的轉錄因子差異分析,長雄野生稻擁有86個家族共計2095個轉錄因子,其中ERF轉錄因子數量最多(857個),其次是bHLH(128個)、NAC(120個)、MYB(119個)和C2H2(116個)。
總結
本研究依托華大序風CycloneSEQ平臺成功組裝了長雄野生稻的端粒到端粒(T2T)的基因組,該基因組包含完整的12條染色體及24個端粒。與已發表的稻屬其他的參考基因組比較發現栽培稻和野生稻之間眾多的基因組結構變異。本研究還對長雄野生稻與稻屬其他物種的基因組進行片段重復基因、NBS-LRR抗性基因和轉錄因子的比較分析。長雄野生稻全基因組組裝的更新成果對高價值的表型性狀關聯基因研究提供了證據基礎,對未來育種和非洲水稻與稻屬進化研究搭建了高價值平臺。
預印文章鏈接:
https://www.biorxiv.org/content/10.1101/2024.09.05.611405v1

 基因測序儀業務 SEQ ALL
基因測序儀業務 SEQ ALL











 核心技術
核心技術 熱門應用
熱門應用 組學研究
組學研究 文檔下載
文檔下載 已發表文章
已發表文章 用戶成功案例
用戶成功案例 在線數據集
在線數據集 多功能中心
多功能中心 客戶中心
客戶中心 在線支持
在線支持 關于智造
關于智造 專題推薦
專題推薦